La plataforma de Andalucía-Biotec Salud pone en marcha un proyecto de innovación para el desarrollo de tecnologías de transcriptómica espacial de resolución celular y análisis bioinformáticos estandarizados con aplicabilidad sanitaria. El proyecto está liderado por el Instituto de Parasitología y Biomedicina López-Neyra del CSIC-Granada, en colaboración con otras instituciones andaluzas.

La unidad de Bioinformática del Instituto de Parasitología y Biomedicina López-Neyra – CSIC, colabora, dentro del Plan Complementario Andalucía-Biotec Salud, en el desarrollo e implementación de herramientas para el diagnóstico de enfermedades y terapias avanzadas. Este proyecto liderado por los investigadores Eduardo Andrés y Javier Martín, está enfocado en la creación de herramientas bioinformáticas con el objetivo de analizar datos de transcriptómica espacial de células individuales.
En la actualidad, las técnicas más novedosas permiten realizar estudios transcriptómicos de cada una de las células que forman un tejido u órgano de interés, lo que recibe el nombre de secuenciación ARN de célula única, (o en inglés single cell RNA Sequencing o scRNASeq). Esta poderosa técnica tiene aplicaciones extensas en diversos campos, como la oncología, la investigación del desarrollo o la inmunología, entre muchos otros.
El uso de scRNAseq no sólo permite el estudio y la comprensión de los distintos procesos celulares tales como comunicación intercelular o respuestas al entorno, sino que, además, permite identificar la heterogeneidad celular propia de tejidos y órganos, y los distintos papeles que ejerce cada célula en situaciones tanto fisiológicas como de enfermedad.
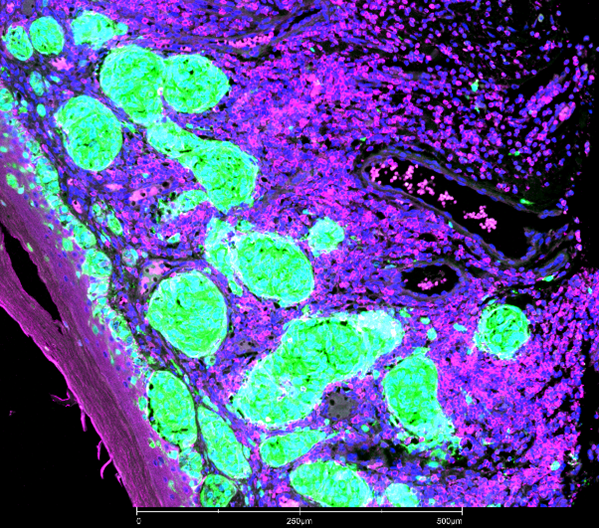
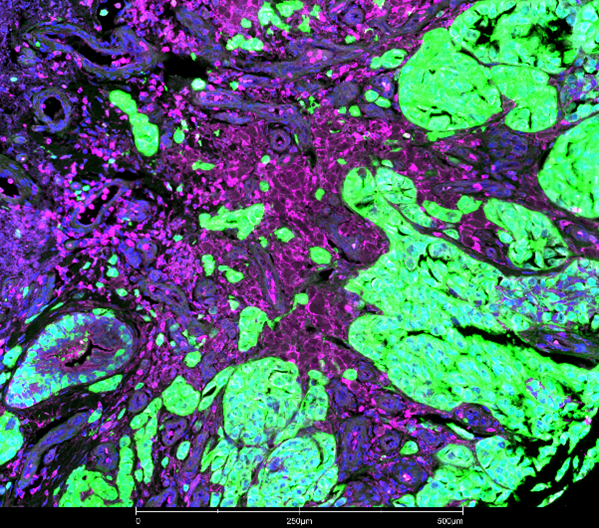
Imágenes de muestras de melanoma humano: las áreas del tumor aparecen en verde, las células inmunitarias en magenta y los núcleos celulares en azul. Fuente: Imágenes generadas por los investigadores de la Universidad de Granada Natividad Martín-Morales, Francisco O’Valle y Pablo Galindo-Moreno.
Sin embargo, el estudio de célula única no tiene en cuenta el entorno celular, así como los diferentes estímulos que recibe cada célula en función de la localización en la que se encuentra. Este nuevo nivel de información representa un punto calve para el diagnóstico y prognosis de las enfermedades. Por ello, el equipo de investigación bioinformática del IPBLN, tienen como objetivo el estudio integrado de muestras de transcriptómica a nivel celular (scRNASeq) junto con su localización en el tejido. Para ello, los investigadores van a trabajar con el sistema de transcriptomica espacial más potente que existe en la actualidad y denominado Stereo-seq: Secuenciación Ómica de Resolución Espacial Mejorada, SpaTial Enhanced REsolution Omics-Sequencing (Stereo-seq).
Esta nueva estrategia innovadora y puntera en el campo de la transcriptómica permite añadir una nueva capa de información que es fundamental en la investigación biomédica y su traslación a la clínica, cumpliendo así un objetivo doble. Por una parte, acercar a la práctica clínica tecnología puntera como es la Stereo-seq, a la vez que la creación de una unidad de bioinformática que actúe como punto de referencia gracias al desarrollo de análisis estandarizados que aseguren resultados robustos.

Integrantes del departamento de Bioinformática del Instituto de Parasitología y Biomedicina López-Neyra (IPBLN). De izquierda a derecha: Laura Terrón, Álvaro Macías, José Luis Ruiz, Eduardo Andrés y Daniel García.
Si quieres tener más información acerca del proyecto, no te pierdas esta entrevista con Eduardo Andrés, uno de sus investigadores principales.












