MITOCANCER
Dirigirse a una enzima de poliglutamilación mitótica: una nueva estrategia para la terapia del cáncer
LINEA DE ACTUACIÓN 2: IMPLEMENTACIÓN Y ANÁLISIS DE BASES DE DATOS EN MEDICINA DE PRECISIÓN
Linea de actuación complementaria: LA3. Plataforma de cribado de fármacos y análisis interacciones fármaco-diana.
Palabras clave: Cáncer de mama, microtúbulo, enzima, polyglutmylacion, terapias
Coordina: Fundació Centre de Regulació Genòmica (CRG)
Participa: Vall Hebron Institut de Recerca (VHIR) (Cataluña), Centro Singular de Investigación en Medicina Molecular e Enfermedades Crónicas (CiMUS) (Galicia)
Persona de contacto: Dr Isabelle Vernos (isabelle.vernos@crg.eu) y Dr Ana Arsenijevic (ana.arsenijevic@crg.eu)
RESUMEN PROYECTO
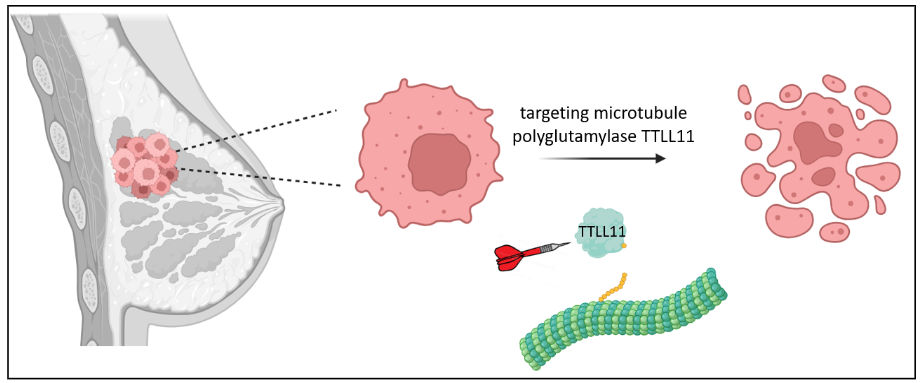
El cáncer de mama es el cáncer más común en las mujeres en todo el mundo y la segunda causa de muerte relacionada con el cáncer. En la mitad de los casos el cáncer reaparece dentro de los primeros cinco años y adquiere resistencia, lo que supone un importante desafío clínico en el manejo de esta enfermedad. Por tanto, se necesitan nuevos objetivos y enfoques para tratar con éxito este tipo de cáncer. Tenemos evidencias preliminares de que una enzima involucrada en la poliglutamilación de los microtúbulos del huso mitótico puede proporcionar una nueva diana interesante como alternativa a los taxanos. Esta enzima está regulada a la baja en tumores humanos, incluido el cáncer de mama, lo que favorece la aneuploidía y CIN (inestabilidad cromosómica), ambas características distintivas del cáncer.
Por otro lado, las células de cáncer mantienen una actividad enzimática mínima de TTLL11 lo que sugiere que necesitan esta actividad para su proliferación y superviviencia. Nuestra hipótesis es que disminuir la actividad de esta enzima por debajo de un cierto umbral en las células cancerosas puede enviarlas a una catástrofe mitótica, desencadenando la formación de micronúcleos y generando respuestas inflamatorias, por lo tanto, la muerte celular y la reducción del tamaño del tumor.
Combinando la experiencia del grupo de la Dra Vernos (Cataluña) en división celular y microtúbulos con la experiencia del Dr. Ramón y Cajal (Cataluña) en cáncer de mama así como con la colaboración con un instituto líder en cribado químico (Dra Loza, Galicia) y análisis genómico (Dr. Carracedo, Galicia), pretendemos obtener una caracterización completa del panorama transcriptómico de tumores de mama de pacientes en las instalaciones del CRG para evaluar la heterogeneidad de los niveles de expresión de la enzima en diferentes tumores y dentro de un mismo tumor. Paralelamente, identificaremos compuestos inhibidores de la enzima y probaremos su potencial para promover la muerte de las células del cáncer de mama.
RESULTADOS DEL PROYECTO
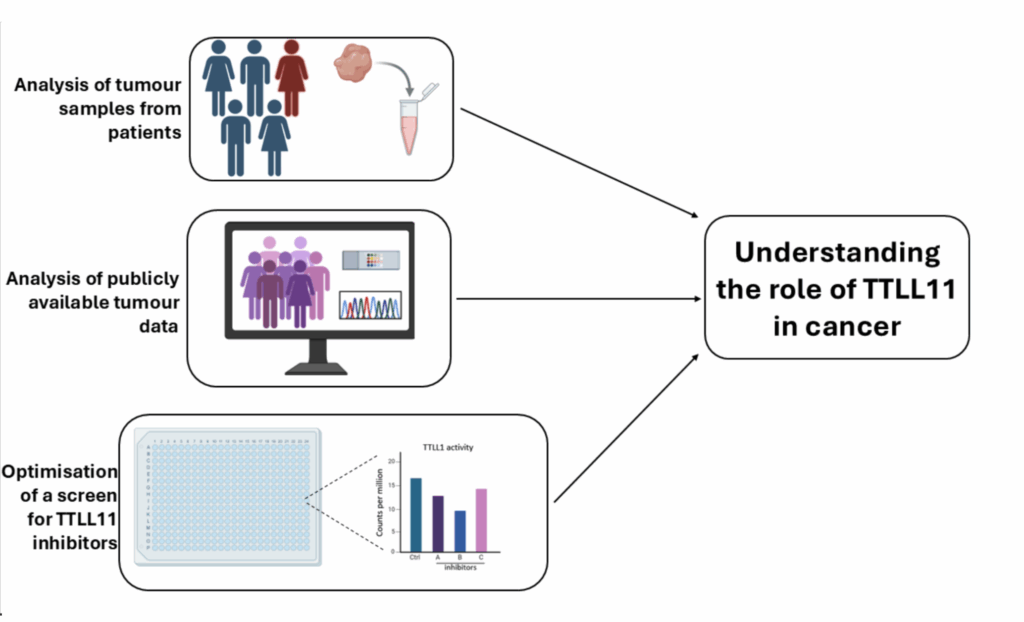
El objetivo inicial de este proyecto fue investigar la heterogeneidad intertumoral e intratumoral de la expresión de TTLL11 en diversos tipos de cáncer y poblaciones celulares mediante secuenciación de ARN unicelular. Sin embargo, observamos que TTLL11 es un gen de baja expresión que se desregula aún más en el cáncer, lo que dificulta su detección con este método. Esta observación coincide con los conjuntos de datos disponibles públicamente, donde nuestros análisis bioinformáticos no lograron identificar una expresión robusta de TTLL11 en ningún tipo de célula.
Reconociendo estas limitaciones, exploramos enfoques alternativos y encontramos evidencia de heterogeneidad en la expresión de TTLL11 en diferentes tipos de tumores. Dado que la qPCR es significativamente más sensible y capaz de detectar TTLL11 de forma fiable tanto en tejidos normales como cancerosos, realizamos un experimento preliminar de RT-qPCR. Nuestro análisis de cáncer de mama y tejido normal compatible de la misma paciente confirmó que los niveles de TTLL11 estaban reducidos en las muestras tumorales. Además, al comparar tumores metastásicos y no metastásicos, observamos una tendencia que sugiere una menor expresión de TTLL11 en subtipos más agresivos.
Para validar aún más estos hallazgos y comprender mejor el mecanismo de acción de TTLL11 en tumores, realizamos una secuenciación masiva de ARN, que confirmó que TTLL11 puede detectarse de forma robusta con este enfoque. Actualmente, estamos a la espera de más muestras de pacientes para realizar análisis estadísticos y determinar las condiciones específicas en las que varía la expresión de TTLL11.
Paralelamente, desarrollamos varios ensayos de poliglutamilación in vitro para medir la actividad de TTLL11, con el objetivo final de optimizarlos para una plataforma de cribado de alto rendimiento que utilizarán nuestros colaboradores en la USC. Para abordar esta limitación, desarrollamos un ensayo de detección alternativo basado en anticuerpos, que parece ser más adecuado para el cribado de alto rendimiento. Este ensayo mejorado está siendo probado actualmente por nuestros colaboradores en la USC.
COORDINA:
PARTICIPA:


