Epigenética en el espectro ELA-DFT: comprensión de la variabilidad fenotípica y nuevos avances en medicina personalizada
LINEA DE ACTUACIÓN 4: DESARROLLO DE MODELOS BIOLÓGICOS PARA CRIBADO Y ESTUDIO DE LA ACTIVIDAD DE MOLÉCULAS TERAPÉUTICAS
Palabras clave: Esclerosis Lateral Amiotrófica, modelos animales, RNA no codificante, epigenética.
Coordina: Universidad de Zaragoza
Participa: Instituto de Investigación Sanitaria Aragón
Persona de contacto: Rosario Osta Pinzolas (osta@unizar.es)
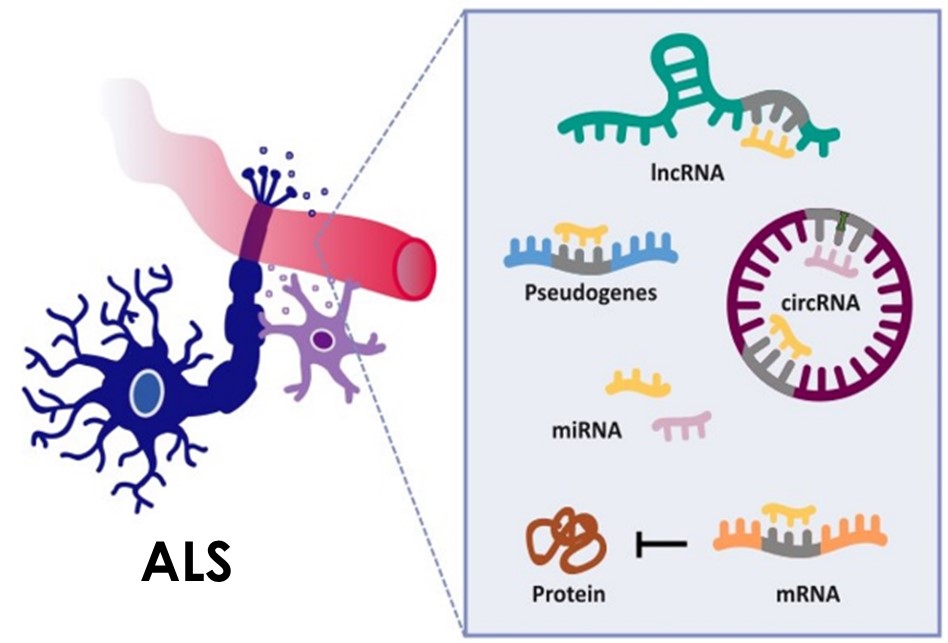
El objetivo general de este proyecto es el hallazgo de nuevos biomarcadores epigenéticos que nos permitan conocer mejor la patogenia y el desarrollo de una medicina personalizada de calidad para los pacientes con Esclerosis Lateral Amiotrófica (ELA) y Demencia Frontotemporal (DFT). Resultados previos de nuestro grupo han demostrado la existencia de variaciones en los microRNAs y en los RNA circulares (circRNAs) en el modelo murino SOD1-G93A de ELA respecto a sus controles sanos. Recientemente, otro mecanismo de procesamiento de RNA, los RNA largos no codificantes (lncRNAs) también se asocian a laenfermedad. El primer objetivo será evaluar las modificaciones que sufren los lncRNAs en el modelo murino SOD1-G93A de ELA en sus estadios iniciales para su posible utilización como marcador diagnóstico (estadios tempranos) y a lo largo de la enfermedad como biomarcador pronóstico, realizando una correlación entre los marcadores modificados y la supervivencia de los animales, resultados que serán posteriormente trasladados a pacientes para comprobar su valor diagnóstico y/o pronóstico de la enfermedad (subproyecto PI21/00286). En el segundo de los objetivos se compararán una batería de biomarcadores epigenéticos ya validados previamente por nuestro consorcio en el modelo de ELA SOD1-G93A en dos fondos genéticos (C57BL6 vs B6SJL), entre los que hemos encontrado una clara diferencia en la supervivencia, para incrementar la robustez de los mismos. Por último, en ambos fondos genéticos, se realizarán estudios comparativos en los patrones de metilación mediante secuenciación del genoma, para corroborar los resultados de los pacientes obtenidos en el subproyecto PI21/00286.
COORDINA:
PARTICIPA:














